アーカイブ
FitzPeaks NaIを用いた放射線測定(核種同定編)
6. 試料データー読み込み
エネルギー校正が完了しましたので、実際の試料の計測を行います。
6-1. 設定変更
エネルギー校正を行った検出器の番号を設定します。
“Set-Up”をクリックして、下記に示す設定項目を変更してください。
今回は検出器“Det_02.cal”ですので、“2”と入力します。
次に、バックグラウンドデーターを指定します。
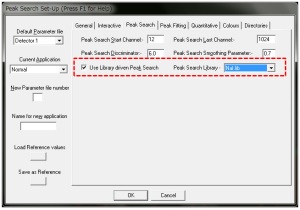
“Peak Fitting”タブ項目に移動し、下図に示す設定項目を編集します。
バックグラウンドデーターは、あらかじめデーターベースに登録しておく必要があります。
6-2. データー読み込み
試料のスペクトルデーターファイルを選択開きます。
すると、収集条件などを入力するウィンドウが表示されますので、必要事項を入力して“OK”ボタンをクリックします。
Detector File: 2が設定されていることを確認してください。
Live Time: 収集時間を入力します。
7. 核種同定(核種ライブラリー編集)
“Analyse”→“Library Peak Search”をクリックします。これは、核種ライブラリーに基づくピークを検出します。
前述の“Peak Search”とは異なるので注意してください。
ピーク検出したら、
“Nuclides”→“Show Nuclides”および
“Nuclides”→“Nuclide ID for Cursor”
をクリックすると、ピークに応じた核種を表示することができます。
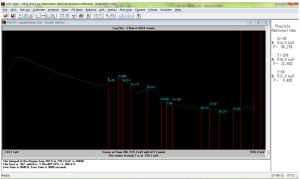
ところが、上図に示すように正しく核種を同定できないことがあります。
原因は、
・エネルギー校正が厳密ではない
・核種ライブラリーが適切ではない
ことが考えられます。
エネルギー校正に関しては、用意できるエネルギー校正用線源に依存します。今回は簡易校正のために十分なエネルギー校正が行えなかったと考えられます。
核種ライブラリーに関しては、デフォルトで指定されているライブラリーに必要な核種が含まれていないためです。
“Nuclides”→“Nuclide List”をクリックすると、ライブラリーに登録されている核種を確認することができます。
このライブラリーでは、Cs-134がリストに含まれていないため、他の核種に同定されてしまっているのが分かります。
そこで、他の核種ライブラリーを指定する方法を説明します。
一度“Analyse”→“Clear Peak List”をクリックしてピークを消去し、
“Set-Up”をクリックして設定を変更します。
“Peak Search”タブ項目の“Use Library driven Peak Search”チェックボックスをONに変更し、核種ライブラリーは“NaI.lib”を選択します。
次に、“Quantitative”タブ項目の“Analysis Library”項目に“NaI.lib”を選択します。
核種ライブラリーを選択したらもう一度
“Analyse”→“Library Peak Search
“Nuclides”→“Show Nuclides”
“Nuclides”→“Nuclide ID for Cursor”
を試行します。
すると、今度はかなり多くのピークを検出しましたが、逆に多すぎるし、適切な核種を同定されていない結果になってしまいました。
下図に示す“Expand”をクリックすると、スペクトル表示を拡大することができます。
“Nuclides”→“Nuclide ID for Cursor”をクリックすると、カーソルで指定したエネルギー付近に予想される核種が表示されます。
上図のカーソルで示すピークは、採取した土壌試料から考えるとCs-134(795.9 keV)と要するのが妥当と思われますが、Co-58(810 keV)と表示されてしまいました。現在多く存在してしまっている核種はCsです。かつ誤検出されたCo-58の半減期は約70日ですので、Co-58が検出されたのは間違えと考えられます。
そこで、誤検出されないように、想定している核種のみのライブラリーデータを作成することにします。
7-1. ライブラリーデータコピー
ライブラリーデータは、FitzPeaks NaIフォルダー下の“Libraries”フォルダー内にあります。
“NaI.lib“ファイルを任意の名前でコピーしてください。
今回は”NaIBasama.lib”というファイルで保存しました。
7-2. ライブラリーデータ読み込み
“File”→“Edit Analysis Library”をクリックすると“”ウィンドウが表示され、ライブラリーデータを編集することができます。
“Open Library”ボタンをクリックして、先ほど作成したライブラリーデータを読み込みます。
“Current Library name”に“NaIBasama.lib”と表示されていることを確認してください。
8. ライブラリーデータ編集
次に、不必要な核種を削除します。
“”ウィンドウ左側のリストに表示されている核種を選択し、“Delete Nuclides”ボタンをクリックして削除します。
今回は、Cs, Kのみ残しました。
最後に“Save Library”ボタンをクリック→“Close”ボタンをクリックして終了します。
9. ライブラリーデータ適応
作成したライブラリーを適応します。
再度“Set-Up”ボタンをクリックし、
“Peak Search”タブ項目の“Use Library driven Peak Search”チェックボックスをONに変更し、核種ライブラリーは“NaIBasama.lib”を選択します。
次に、“Quantitative”タブ項目の“Analysis Library”項目に“NaIBasama.lib”を選択します。
核種ライブラリーを選択したらもう一度
“Analyse”→“Library Peak Search
“Nuclides”→“Show Nuclides”
“Nuclides”→“Nuclide ID for Cursor”
を試行します。
下図に結果を示します。
正しく核種が認識されるようになりました。
核種同定については以上です。
次は、ピーク面積解析を行う方法をまとめたいと思います。